复旦大学生命科学学院李继喜团队在抗病毒免疫应答研究领域取得重要进展
近日,复旦大学生命科学学院教授李继喜团队在病毒-宿主天然免疫应答的结构基础和作用机理研究领域取得重要进展。6月9日,研究论文以《人源RNA解旋酶DDX21结合和解旋RNA及在抗病毒信号激活中的结构基础》(“Structural Basis of Human Helicase DDX21 in RNA Binding, Unwinding, and Antiviral Signal Activation)为题发表于《尖端科学》(Advanced Science)杂志。
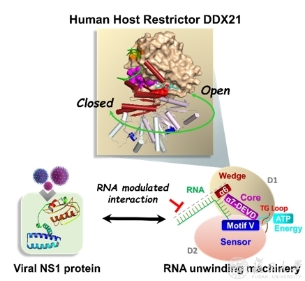
人源RNA解旋酶DDX21的解旋机理及RNA介导的DDX21-NS1相互作用
季节性流感病毒在全球范围内每年会导致29-65万例死亡和300-500万例严重感染。而同属于单链RNA病毒的新型冠状病毒在2020年更是肆虐蔓延迅速发展成为了全球性的疫情,造成灾难性的损失。研究RNA类病毒的致病机理,对相关新药研发包括疫苗的研制都具有重要意义。DDX21是一类重要的DEAD-box RNA解旋酶,兼具ATP依赖的RNA解旋酶活性和RNA折叠酶活性。DDX21在细胞生命周期中扮演多种重要角色,调控多种肿瘤组织的发生发展;还被报道与多种RNA病毒相互作用,通过形成DDX1-DDX21-DHX36-TRIF复合物来作为一类重要的病毒核酸传感器。作为宿主的限制性调节因子,DDX21与病毒PB1蛋白结合从而抑制病毒聚合酶的组装,抵抗流感病毒A的复制和合成。在病毒感染后期,病毒NS1蛋白可以与DDX21结合而释放PB1,从而逃避机体的天然防御机制。
在该项研究中,李继喜团队获得了DDX21的多套高分辨率晶体结构,包括RNA解旋的初始状态 (DDX21-apo,3.1Å分辨率),结合AMPPNP和ssRNA的解旋后状态 (DDX21-AMPPNP-ssRNA,2.24Å分辨率), 以及结合ADP的水解后状态 (DDX21-ADP,1.8Å分辨率),揭示了在整个解旋循环中RNA解旋酶核心的两个保守结构域在开放和闭合之间的连续构象转换,并阐述了包含楔形螺旋 (基序Ic),传感器 (基序V) 和DExD-box基序核心的精巧解旋机器,以及全长蛋白的三分子协同作用解旋机理。此外,结合共表达、共纯化以及小RNA测序的结果,研究者发现小RNA、DDX21和病毒的NS1蛋白可协同组装成复合物,并且NS1可通过小RNA介导来抑制DDX21的ATP酶水解活性和RNA解旋酶活性;更重要的是,病毒感染细胞中NS1解除宿主DDX21的抑制作用也是通过小RNA介导的。这些研究为DDX21的RNA识别和解旋机制提供了结构基础,并为病毒-宿主相互作用界面提供了新的视角,这将为开发与流感病毒感染相关的药物提供新的作用靶标和治疗方法奠定基础。
生命科学学院博士研究生陈子珺为论文第一作者,李继喜为论文通讯作者。该研究得到了科技部国家重点研发计划、国家自然科学基金委以及上海市科委国际合作项目的资助。

人源RNA解旋酶DDX21的解旋机理及RNA介导的DDX21-NS1相互作用
季节性流感病毒在全球范围内每年会导致29-65万例死亡和300-500万例严重感染。而同属于单链RNA病毒的新型冠状病毒在2020年更是肆虐蔓延迅速发展成为了全球性的疫情,造成灾难性的损失。研究RNA类病毒的致病机理,对相关新药研发包括疫苗的研制都具有重要意义。DDX21是一类重要的DEAD-box RNA解旋酶,兼具ATP依赖的RNA解旋酶活性和RNA折叠酶活性。DDX21在细胞生命周期中扮演多种重要角色,调控多种肿瘤组织的发生发展;还被报道与多种RNA病毒相互作用,通过形成DDX1-DDX21-DHX36-TRIF复合物来作为一类重要的病毒核酸传感器。作为宿主的限制性调节因子,DDX21与病毒PB1蛋白结合从而抑制病毒聚合酶的组装,抵抗流感病毒A的复制和合成。在病毒感染后期,病毒NS1蛋白可以与DDX21结合而释放PB1,从而逃避机体的天然防御机制。
在该项研究中,李继喜团队获得了DDX21的多套高分辨率晶体结构,包括RNA解旋的初始状态 (DDX21-apo,3.1Å分辨率),结合AMPPNP和ssRNA的解旋后状态 (DDX21-AMPPNP-ssRNA,2.24Å分辨率), 以及结合ADP的水解后状态 (DDX21-ADP,1.8Å分辨率),揭示了在整个解旋循环中RNA解旋酶核心的两个保守结构域在开放和闭合之间的连续构象转换,并阐述了包含楔形螺旋 (基序Ic),传感器 (基序V) 和DExD-box基序核心的精巧解旋机器,以及全长蛋白的三分子协同作用解旋机理。此外,结合共表达、共纯化以及小RNA测序的结果,研究者发现小RNA、DDX21和病毒的NS1蛋白可协同组装成复合物,并且NS1可通过小RNA介导来抑制DDX21的ATP酶水解活性和RNA解旋酶活性;更重要的是,病毒感染细胞中NS1解除宿主DDX21的抑制作用也是通过小RNA介导的。这些研究为DDX21的RNA识别和解旋机制提供了结构基础,并为病毒-宿主相互作用界面提供了新的视角,这将为开发与流感病毒感染相关的药物提供新的作用靶标和治疗方法奠定基础。
生命科学学院博士研究生陈子珺为论文第一作者,李继喜为论文通讯作者。该研究得到了科技部国家重点研发计划、国家自然科学基金委以及上海市科委国际合作项目的资助。
我要收藏
个赞











评论排行